Traduction
Tutoriel Python - sommaire
Tutoriel Python - recherche
L'auteur : Patrick Darcheville
 Vous pouvez me contacter via Facebook pour questions & suggestions :
Page Facebook relative à mon site
Vous pouvez me contacter via Facebook pour questions & suggestions :
Page Facebook relative à mon site
 Vous pouvez me contacter via Facebook pour questions & suggestions :
Page Facebook relative à mon site
Vous pouvez me contacter via Facebook pour questions & suggestions :
Page Facebook relative à mon site
 La galaxie Python propose un SGBDR (Système de Gestion de Base de Données Relationnelles) très intéressant : SQLite.
La galaxie Python propose un SGBDR (Système de Gestion de Base de Données Relationnelles) très intéressant : SQLite.
Une base de données de type SGBDR peut être manipulée par des requêtes écrites en SQL (Structured Query Language).
 Richard Hipp, le créateur de SQLite, commence la conception de ce moteur de
SGBD en 2000 suite à une commande de la défense US ; SQLite devant équiper des missiles guidés.
Richard Hipp, le créateur de SQLite, commence la conception de ce moteur de
SGBD en 2000 suite à une commande de la défense US ; SQLite devant équiper des missiles guidés.
Une base de données a un nom et comprend une ou plusieurs tables. Elle comprend aussi éventuellement des vues.
Un table c'est comme un tableau : des lignes et des colonnes. Les colonnes sont aussi appelée champs ("fields") et les lignes sont
aussi appelées enregistrements ("records").
A l'intersection d'une colonne et d'une ligne se trouve une cellule contenant une donnée.
Chaque ligne décrit un individu (au sens statistique du terme) : un salarié, un client, un contact, un article du catalogue, un patient,
etc.
Dans un SGBDR tous les individus sont donc décrits de la même façon !
Nous devons gérer les espèces animales d'un milieu marin.
Une espèce a un nom et appartient à un groupe (ou taxon). Je rappelle qu'un taxon est un groupe d'animaux "cousins" (qui ont un ancêtre commun).
La base de données se nommera "animaux.db". Elle comprendra donc deux tables :
Ce que doit contenir la table taxons (extrait) :
Une ligne (ou enregistrement) par taxon.
Ce que doit contenir la table "especes" (extrait) :
1 homard deca 2 orange de mer epon 3 fesses d'éléphant epon 4 crevette grise deca 5 bouquet deca 6 étrille deca ...
Remarque : une ligne (ou enregistrement ou "record") par espèce.
Dans toute table une colonne (et une seule) doit faire fonction de clé primaire.
Il ne peut y avoir de doublons pour cette colonne. Il y aura automatiquement création d'un index sur cette colonne. Donc la recherche
d'une ligne sera extrèment rapide : recherche indexée.
Il y a une colonne commune entre les deux tables ; elle porte d'ailleurs le même nom : "code" (mais ce n'est pas obligatoire).
Cette colonne commune aux deux tables est appelé "champ de jointure". En effet elle permet de mettre en relation les deux tables.
Ainsi à chaque enregistrement de "espèces" on pourra associer une ligne (et une seule) de "taxons".
A contrario pour chaque ligne de "taxons" on peut associer 0 à N lignes de "especes".
Il s'agit donc ici d'une jointure "1 à N".
On dit que "taxons" est table mère par rapport à "espèces" ET que "espèces" est table fille par rapport à "taxons".
En effet une mère peut avoir plusieurs filles mais une fille n'a qu'une mère.
Le langage SQL peut paraitre difficile pour un débutant. Aussi nous allons créer et remplir les tables de notre base SQLite avec un logiciel
intitulé DB Brower for SQLliste.
Cette interface permet d'administrer une base SQLite sans connaitre SQL. Ce logiciel est l'équivalent de PHPMyAdmin pour MySQL.
En fait cette interface génère du SQL à partir de tous les renseignements que vous lui fournissez dans le cadre de grilles.
Disons qu'avec DB browser vous allez découvrir en douceur le SQL.
Il suffit de se rendre dans la page de téléchargement du site sqlitebrower.org : https://sqlitebrowser.org/dl/
Nous allons employer cette interface pour créer la base de données "animaux.db" et les différentes tables.
Il faut maintenant créer chacune des deux tables (définir leur structure) puis leur ajouter des données (insérer des enregistrements).
Il faut donc générer pour chaque table deux requêtes SQL : une de type CREATE TABLE ... et plusieurs de type INSERT INTO ...
Cliquez sur "Créer une table".
Vous remplissez la fenêtre qui s'affiche comme suit :
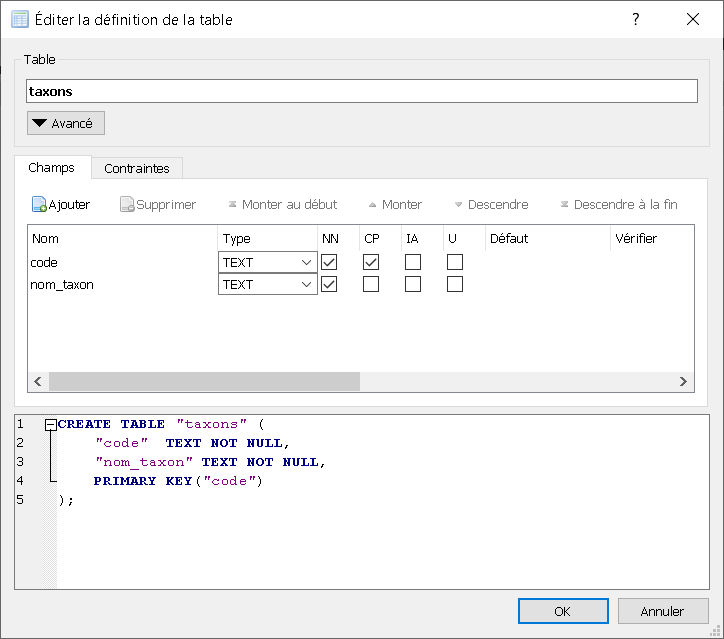
Notez que la grille est légendée "éditer la définition de la table".
Différence et U et CP : dans les deux cas les doublons pour cette colonne sont impossible. Mais avec CP il y a création d'un index. Ce qui n'est pas le cas avec U.
Observons attentivement le code SQL généré :
Pour la table "taxons" il n'y a que deux colonnes à définir.
Les deux colonnes sont de type "text". La première est clé primaire (pour identifier de manière unique une ligne).
Mais comme le type est TEXT, l'autoincrémentation est bien sûr impossible.
La table "taxons" existe désormais (a une structure) mais est vide de toute données. Il faut désormais y ajouter des lignes (ou enregistrements).
Sélectionnez la table "taxons" à partir de la fenêtre "parcourir les donnees"
puis cliquez sur l'icône "insérer un nouvel enregistrement dans la table" : vous pointez alors la fin de table. il suffit de remplir la ligne
vierge du tableau.
Recommencez la procédure autant de fois que nécessaire.
Après avoir saisi plusieurs lignes dans la table "taxons" vous pouvez trier par valeurs croissantes/décroissantes les noms des taxons. Il suffit à partir du contenu sous forme d'un tableau (fenêtre "parcourir les données) de cliquer sur l'étiquette de colonne.
Attention le code SQL de création de la table "especes" est plus complexe que pour la table "taxons".
Il y a effet une contrainte d'intégrité référentielle : on ne doit pouvoir saisir dans la colonne especes.code
qu'une valeur qui existe déjà dans la colonne taxons.code. Il faut donc définir la colonne especes.code comme clé étrangère
par rapport à taxons.code.
CREATE TABLE "especes" ( "id" INTEGER NOT NULL,
"nom_espece" TEXT NOT NULL, "code" TEXT NOT NULL,
PRIMARY KEY("id" AUTOINCREMENT),
FOREIGN KEY("code") REFERENCES "taxons"("code") )
Intéressons nous à la clause
FOREIGN KEY("code") REFERENCES "taxons"("code") :
la colonne "code" de la table 'especes" est clé étrangère par rapport à la colonne
"taxons.code".
Ce qui veut dire qu'on ne peut saisir dans la première qu'une valeur qui existe déjà dans la seconde.
À partir de la fenêtre "parcourir les données" sélectionnez "especes" dans la liste déroulante "table" puis cliquez
sur l'icône "insérer un nouvel enregistement".
Notez que c'est désormais une grille de saisie qui apparait (et non pas un tableau du fait de l'intégrité référentielle).
Cette grille est légendée : "ajouter un enregistrement".
En haut de la grille et pour chaque champ, il faut saisir la valeur (sauf pour le champ ID qui se remplie automatiquement).
Pour "nom_espece" saisissez "cristatelle" et pour "code" saisissez "bryo" (comme bryozoaires) : un message d'erreur apparait :
"Foreign key constraint failed ..." . La requête échoue ! En effet vous n'avez pas pas respecté l'intégrité référentielle ; la valeur "bryo"
n'existe pas encore dans "taxons.code". Dans un langage imagé on dirait : "il ne peut y avoir de fille sans une mère".
Saisissez alors "mérou" & "osse".
Puis observez le code SQL que vous avez généré (affiché en bas de la grille):
INSERT INTO "main"."especes"
("nom_espece", "code")
VALUES ('mérou', 'osse');
Cette fois l'insertion est autorisée puisque la valeur "osse" (poissons osseux) existe dans la colonne "taxons.code".
Dons si vous voulez ajouter des espèces appartenant au taxon des "bryozoaires" il faudra d'abord créer ce taxon dans la table "taxons" puis saisir les espèces de ce groupe dans la table 'especes".
Après avoir saisi plusieurs lignes dans la table "especes" vous pouvez trier par valeurs croissantes/décroissantes les codes et les noms d'espèces. Il suffit à partir du contenu sous forme d'un tableau (fenêtre "parcourir les données) de cliquer sur l'étiquette de colonne ad hoc.
Les requêtes sélection ne modifient pas les données (à la différence des requêtes action telles INSERT INTO ...DELETE FROM ... DROP ...). Elles se contentent d'afficher des données extraites d'une ou plusieurs tables avec éventuellement tri, limite et filtage.
Dans l'outil DB Browser vous êtes obligé de tapez les requêtes sélection en SQL (il n'y a pas d'interface qui génère cette requête).
Cliquez sur "exécutez le SQL" ; tapez le code de la requête sélection puis cliquez sur la flèche bleu orientée vers la droite.
De la table "especes" : SELECT * FROM especes
De la table "taxons" : SELECT * FROM taxons
* veut dire "afficher toutes les colonnes" de la table
Colonne "nom_espece" & "code" : SELECT nom_espece, code FROM especes
Donc la colonne ID n'est pas affichée.
Il est d'usage pour les mots réservés du langage SQL soient écrits en majuscules et que les
noms des colonnes et des tables soit délimitées par des guillemets (simples ou doubles).
Mais le fait d'omettre les guillemets et d'écrire les mots réservées en minuscules ne constituent des erreurs de syntaxe.
Ainsi au lieu d'écrire SELECT 'nom_espece' FROM 'especes' vous pouvez écrire tout simplement select nom_espece from especes
Déns les deux cas la requête sera exécutée avec succès.
Il s'agit d'une requête SELECT avec la clause LIMIT suivi de 1 ou 2 arguments.
SELECT * FROM especes LIMIT 10 : affichage des 10 premières lignes
SELECT * FROM especes LIMIT 10,30 : affichage de 22 lignes (de la 10ième à la 30ième incluses)
Afficher seulement les décapodes (code ="deca") :
SELECT nom_espece, code FROM especes WHERE code ="deca"
Afficher seulement les poissons osseux (code ="osse") :
SELECT * FROM especes WHERE code ="osse"
Afficher poissons osseux et décapodes :
SELECT * FROM especes WHERE code ="osse" or code="deca"
La condition de filtrage devient complexe.
SELECT * FROM especes WHERE nom_espece LIKE "h%"
On affiche seulement les espèces dont le nom commence par la lettre "h" (minuscule ou majuscule car LIKE est insensible à la casse).
LIKE s'associe à "%" qui remplace n'importe qu'elle chaîne de caractères et "_" qui remplacement un seul et unique caractère.
Vous pouvez écrire : SELECT * FROM especes WHERE nom_espece LIKE "%de mer%".
Cette requête retourne les lignes dont le champ "nom_espece" contient l'occurence "de mer" .
Dans le chapitre suivant vous verrez que le critère de filtrage peut être saisi par l'utilisateur : requêtes paramétrées.
SELECT * FROM especes WHERE id BETWEEN '1' AND '10'
On affiche uniquement les enregistrements dont l'id est compris entre 1 et 10, donc le début de table
SELECT * FROM especes WHERE nom_espece BETWEEN 'a%' AND 'd%' order by nom_espece
On affiche les espèces dont la première lettre du nom est compris entre a et d.
SELECT nom_espece, code FROM especes ORDER BY code ASC
Dans la liste retournée, les lignes sont triées par valeurs croissantes pour la colonne code.
L'argument ASC (ascending) est facultatif : valeur par défaut est ASC.
SELECT nom_espece, code FROM especes ORDER BY code DESC
Dans la liste retournée, les lignes sont triées par valeurs décroissantes pour la colonne code.
L'argument DESC est obligatoire.
SELECT * FROM especes WHERE code ="osse" ORDER BY nom_espece
Dans la liste retournée, seuls les poissons osseux figurent et ils sont ordonnées par nom.
Attention la clause WHERE doit précéder la clause ORDER.
L'ordre physique des enregistrements dans la table n'est pas modifié par les tris et filtrages des requêtes SELECT
Nous voulons associer à chaque ligne de "especes" un enregistement (et un seul) de "taxons" puisque chaque espèce appartient à un taxon
et un seul.
Entre "especes" et "taxons" nous avons un champ commun qui porte le même nom dans les deux tables : "code".
Nous pouvons donc poser une jointure entre ces deux tables.
SELECT * FROM especes, taxons WHERE especes.code = taxons.code
La requête retourne autant de lignes que d'enregistements dans la table "especes".
Pour lever toute ambiguïté il faut employer les noms complets des champs de jointure (nomtable.nomColonne) : "especes.code" d'une part
et "taxons.code" d'autre part.
D'ailleurs essayez a requête SELECT * FROM especes, taxons WHERE code = code : vous aurez une erreur !
Désigner des champs de jointure par le même nom (ici "code") est une maladresse.
Mais c'est une maladresse délibérée de ma part ; il faut parfois montrer ce qu'il ne faut pas faire.
Il aurait été préférable de désigner le champ "taxons.code" par "t_code" et le champ "especes.code" par "e_code" donc
préfixer les champs de jointure.
Le rendu (extrait) :
1 homard deca deca décapodes 4 crevette grise deca deca décapodes 5 bouquet deca deca décapodes 6 étrille deca deca décapodes 7 tourteau deca deca décapodes 8 araignée mer deca deca décapodes 9 bulot gast gast gastéropodes ...
Pour chaque ligne on a deux fois la même info (deux fois le code du taxon).
SELECT nom_espece, taxons.code, nom_taxon FROM especes, taxons WHERE especes.code = taxons.code
Cette fois on affiche que les champs précisés après le mot SELECT : le code du taxon n'est affiché qu'une fois !
Pour lever toute ambiguïté il faut écrire "taxons.code" OU "especes.code".
Le rendu (extrait) :
homard deca décapodes crevette grise deca décapodes bouquet deca décapodes étrille deca décapodes tourteau deca décapodes araignée de mer deca décapodes bulot gast gastéropodes ...
Nous avons utilisé le mot clé WHERE pour poser une jointure entre deux tables et si nous voulions aussi effectuer un filtrage en plus on serait bien ennuyé ... on ne peut pas avoir deux clauses WHERE dans la même requête.
Il faut alors utiliser la clause INNER JOIN pour poser une jointure entre deux tables.
Le code SQL devient alors :
SELECT nom_espece, taxons.code as code, nom_taxon FROM especes INNER JOIN taxons ON especes.code = taxons.code
Notez l'emploi d'un alias pour une colonne sélectionnée : taxons.code as code.
Nous allons maintenant sauvegarder cette requête sous forme d'une vue.
A partir de la fenêtre "Exécuter le SQL" cliquez sur l'icône légendé "Enregistrer la vue des résultats". Nommons la vue "vue1".
Donc une vue c'est tout simplement une requête sélection enregistrée.
Je vous propose maintenant une requête avec un code SQL assez lourd ...
SELECT nom_espece, taxons.code as code, nom_taxon FROM especes INNER JOIN taxons ON especes.code = taxons.code
WHERE nom_espece BETWEEN "b%" AND "m%" ORDER BY nom_espece
Le rendu (totalité) :
nom_espece code nom_taxon balane cirr cirripèdes bouquet deca décapodes bulot gast gastéropodes calmar ceph céphalopodes cigale ... deca décapodes cione asci ascidies comatule crin crinoïdes coque biva bivalves crabe .. deca décapodes crevette deca décapodes crépidule gast gastéropodes dugong sire siréniens fraise ... asci ascidies homard deca décapodes huitre biva bivalves lamantin sire siréniens langouste deca décapodes langoustine deca décapodes lion de mer pinn pinnipèdes
Cette requête affiche les espèces dont le nom commence par les lettres "b" à "m" ; espèces classées par odre alphabétique.
Il est préférable, vu sa complexité, de la sauvegarder sous forme d'une vue ; je la nomme "vue2".
Parmi les requêtes SELECT il y a les requêtes statistiques.
Exemples :
Ces requêtes ne retournent qu'une ligne affichant un entier (le nombre de lignes de la source).
Dans la table "espèces" et pour chaque valeur de code il y a plusieurs lignes (plusieurs espèces par taxon).
SELECT code, COUNT(*) FROM especes GROUP BY code
Le rendu (extrait) :
... biva 6 cart 2 ceph 3 cirr 2 crin 1 deca 11 ...
Nous pouvons dire qu'il y a 4 biva(lves), 2 cart(ilagineux), 3 ceph(alopodes), etc.
Cette requête statistique comprend autant de lignes que de groupes ; requête statistique par groupe.
Ces deux requêtes retournent respectivement la valeur maxi et mini dans la colonne ID.
Remarque : suite à des suppressions de lignes, il peut y avoir des trous dans la numérotation : un
enregistrement ne changera jamais d'ID même si des lignes qui le précédaient ont été supprimées.
a titre d'exemple la valeur maxi de ID dans ma table "especes" est de 56 alors que le nombre de lignes est 40.
En effet de nombreuses lignes ont été supprimées dans cette table.
Pour supprimer tout ou partie des enregistrements dans une table il faut exécuter une requête "DELETE FROM nomtable".
Une requête DELETE FROM nomTable sans condition supprime toutes les lignes.
Par contre une requête DELETE FROM nomTable WHERE ... supprime seulement les lignes vérifiant la condition.
Cependant dès qu'une table est soumise à une contrainte d'intégrité référentielle ça devient un peu plus compliqué ... Lisez le paragraphe qui suit.
Mais attention dans le cadre de l'intégrité référentielle on ne peut pas supprimer "une mère tant qu'elle a des filles".
Par contre rien n'interdit de supprimer des lignes dans la table "fille".
Essayez la commande : DELETE FROM especes WHERE code ='epon'. C'est un succès !
Compte tenu de toutes les modifications apportées la base "animaux.db" se présente comme suit :

Cet onglet affiche le code de création des tables mais aussi des vues.
La table "sqlite_sequence" est créée par le logiciel SQLite3. Elle est utilisée lorsqu'une colonne dans une table est AUTOINCREMENT .
Dans la base "animaux.db" aucun champ ne contient de date & heure.
Or c'est une problèmatique fréquente dans le cadre d'un SGBD. Ainsi il faut stocker des dates de naissance, des dates d'entrée, des dates de
création de document, etc.
Je ne peux terminer ce chapitre sur SQLite sans vous montrer comment manipuler les dates & heures avec ce SGBD.
Plutôt que de créer une nouvelle base je vais rajouter provisoirement deux tables : "posts" & "eleves" dans la base
"animaux.db".
Concernant les types de champs, SQLite propose : integer, text, real, numérique mais pas vraiement de type date ...
Alors comment faire ???
Dans SQLite la problématique est différente selon qu'il s'agit de l'instant présent ou d'une date historique.
Par exemple dans le cadre d'un blog il faut afficher la date de création de chaque "post".
CREATE TABLE "posts" ( "id" INTEGER,
"date_creation" INTEGER NOT NULL DEFAULT CURRENT_TIMESTAMP,
"contenu" TEXT NOT NULL,
PRIMARY KEY("id") )
Notez la clause : "date_creation" INTEGER NOT NULL DEFAULT CURRENT_TIMESTAMP
Pour définir cette table et plus précisément le champ "date_creation" il faut remplir la colonne "défaut" dans la grille avec
la valeur "CURRENT_TIMESTAMP".
Ce qui veut dire que ce champ sera rempli automatique lors de la création de l'enregistrement avec le timestamp à ce moment précis.
Le timestamp est le nombre de secondes écoulées depuis le 1er janvier 1970 et l'instant présent.
1 2023-12-01 18:40:17 Texte de la première ligne 2 2023-12-01 18:40:42 Texte de la deuxième ligne 3 2023-12-01 18:41:14 Texte de la troisième ligne
Heureusement un champ rempli avec des timestamps s'affiche sous forme d'une info lisible pour un francophone :
date et heure (AAAA-MM-JJ HH:MM:SS)
Vous constatez que les lignes ont été créées le même jour (1er décembre 2023).
Je vous propose :
SELECT * FROM posts where date_creation < date('2024-01-01')
Requête qui retourne trois lignes, bien sûr.
Autre solution :
SELECT * FROM posts where year(date_creation) < 2024
Emploi de la fonction year() : pas d'erreur : affichage de trois lignes !
Cette fois le remplissage automatique du champ n'est pas envisageable puisque la date ne correspond pas à l'instant présent.
Il faut créer un champ de type text et saisir toutes les dates au même format : aaaa-mm-jj (avec des tirets)
CREATE TABLE "eleves" (
"id" INTEGER,
"nom" TEXT NOT NULL,
"prenom" TEXT NOT NULL,
"sexe" integer default 1,
"date_naissance" TEXT NOT NULL,
PRIMARY KEY("id" AUTOINCREMENT)
)
Excusez moi mesdames, je suis très macho, la valeur par défaut pour le champ "sexe" est 1 (masculin)
Saisir quatre individus dans cette table : un né en 1979, un autre en 1980, un troisième né en 1981 et un quatrième en 1982. Tous sont nés le 1er janvier ; deux femmes et deux hommes.
1 Dupont louis 1 1979-01-01 2 Dubois hélène 2 1980-01-01 3 Durant julien 1 1981-01-01 4 Dumond pauline 2 1982-01-01
SELECT * FROM eleves WHERE date_naissance < date('1981-01-01')
Cette requête fonctionne : emploi de la fonction date argumentée avec une date au format (aaaa-mm-jj).
SELECT * FROM eleves WHERE year(date_naissance) = 1982
Cette fois ça ne marche pas car le champ date_naissance est de type text donc je ne peux employer
la fonction year().
La commande "DELETE FROM nomTable" sans clause WHERE permet de vider une table de tout contenu ; par contre sa définition est sauvegardée. Donc vous videz la table mais ne la supprimer pas.
Mais "eleves" & "posts" sont toujours dans la liste des tables et on retrouve toujours leur définition (create table ...) dans la structure de la base de données.
Il ne faut pas confondre vidage de table avec suppression de table !
Il faut maintenant supprimer les tables provisoires "posts" & "eleves" de la base ; elles nous ont été utiles pour nos essais. Mais désormais nous n'en n'avons plus besoin.
Exécutez les commandes : DROP TABLE eleves & DROP TABLE posts
Les deux tables sont bien supprimées : elles ne sont plus dans la liste des tables et leur définition n'apparait plus.